Comment reconstituer l’évolution des espèces avec plus de précision que jamais ? Nicolas Salamin et ses collègues repoussent les limites de la biologie évolutive grâce à l’intelligence artificielle. En s’affranchissant des hypothèses simplificatrices des modèles traditionnels, leur modèle « phyloRNN » utilise le deep learning pour estimer les paramètres de l’évolution moléculaire directement à partir des séquences d’ADN. Une avancée majeure qui ouvre de nouvelles perspectives pour la recherche sur la biodiversité.
Imaginez essayer de reconstruire l’histoire évolutive de la vie en analysant les séquences d’ADN se trouvant dans les génomes de chaque individu. Les scientifiques ont traditionnellement utilisé des modèles mathématiques pour le faire, mais ces modèles reposent souvent sur la simplification d’hypothèses qui pourraient ne pas saisir pleinement la complexité de l’évolution. Nicolas Salamin et ses collègues Daniele Silvestro de l’ETHZ et Thibault Latrille de l’UNIL ont dévelopé une nouvelle approche qui utilise la puissance de l’intelligence artificielle, en particulier le Deep Learning, pour relever ces défis.
Ils ont développé un nouveau modèle d’intelligence artificielle, appelé phyloRNN (github.com/phylornn/phylornn), conçu pour analyser directement les alignements multiples des séquences d’ADN et estimer les paramètres cruciaux de l’évolution moléculaire, tels que la vitesse à laquelle différentes parties du génome changent et la divergence globale qui s’est produite, le tout sans avoir besoin d’un arbre évolutif préexistant. Le développement de phyloRNN implique une nouvelle stratégie en couplant des simulations numériques de l’évolution du génome avec un modèle de Deep Learning supervisé (cf figure).
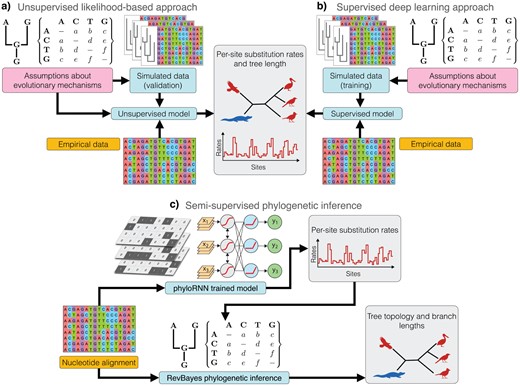
Essentiellement, ils ont créé de grandes quantités de données synthétiques qui imitaient des scénarios du monde réel, y compris des modèles complexes de variation de taux qui sont difficiles à modéliser avec des méthodes mathématiques traditionnelles. Ces données simulées ont ensuite été utilisées pour entrainer le modèle phyloRNN, lui permettant d’apprendre les relations complexes entre les modèles de séquence d’ADN et les processus évolutifs sous-jacents.
Les prédictions du modèle phyloRNN sur les taux d’évolution se sont révélées aussi précises et, dans de nombreux cas significativement plus précises que celles obtenues à partir de méthodes traditionnelles, en particulier lorsqu’il s’agit de scénarios évolutifs plus complexes. Les chercheurs ne se sont pas arrêtés là. Ils ont ensuite montré comment ces estimations alimentées par l’IA pourraient être réintégrées dans les méthodes phylogénétiques traditionnelles pour améliorer la précision de la reconstruction des arbres évolutifs. En utilisant les taux évolutifs par site prédits par phyloRNN dans un cadre bayésien, ils ont constaté que l’exactitude de l’inférence phylogénétique, en particulier les longueurs des branches estimées, était considérablement améliorée.
Cette approche semi-supervisée innovante, combinant les forces de l’apprentissage en profondeur pour l’estimation des taux avec la rigueur de l’inférence probabiliste pour la construction d’arbres, suggère un avenir prometteur pour l’analyse phylogénétique, permettant d’incorporer des modèles d’évolution plus flexibles et réalistes. Ces recherches vont permettre d’ouvrir la voie à de futurs progrès et à des efforts de collaboration dans les domaines de la biologie computationnelle, de l’informatique et de la recherche en évolution. Le potentiel du Deep Learning en phylogénie inspire d’autres innovations et explorations dans ce domaine interdisciplinaire, contribuant à une compréhension plus approfondie de la dynamique et des mécanismes influençant l’évolution des espèces et de la biodiversité.
Le Professeur Nicolas Salamin est biologiste et intègre la modélisation et l’intelligence artificielle dans ses recherches pour comprendre les mécanismes conduisants à l’évolution des espèces et de la biodiversité.
Faculté de biologie et de médecine
Modeling, Deep Learning, Evolutionary genomics

